如何生成PSSM矩阵(本教程摘自钱昱磬同学的CSDN博客)
1.安装psiblast软件
1.1这里提供了windows下了64位版本:ncbi-blast-2.3.0+-win64.exe(最新版的win和linux版可去官网下载)。
1.2下载后是一个exe文件,直接双击安装,注意安装路径不要在带有空格的文件夹下,其他下一步就可以了,没有特别注意的地方。
1.3添加环境变量,如下图所实例,将bin文件夹的路径添加。环境变量创建完成后,在cmd中输入blastn -version,没有报错就说明成功了。
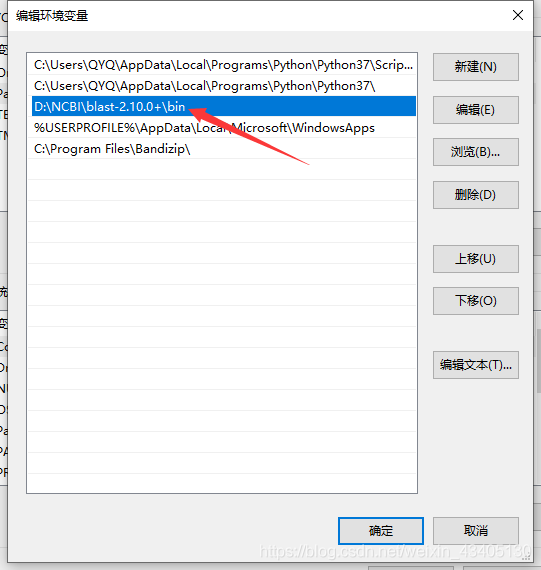
2.添加数据库
2.1 在bin文件夹的同一路径下(不是在bin文件夹下)创建创建db文件夹,将已经编译好的数据库(由于非冗余数据库有20多G,故这里生产了一个可以满足常规需要的小型db)解压缩放入db文件夹内。
3.使用
在这里举个例子:在cmd中输入
psiblast -in_msa D:\NCBI\input\test.fasta -db D:\NCBI\blast-2.3.0+\db\uniprot-all.fasta -comp_based_stats 1 -inclusion_ethresh 0.001 -num_iterations 3 -out_ascii_pssm D:\NCBI\output\1.pssm
-in_msa后面是test.fasta的文件路径,test.fasta文件内有是由头部和序列两个部分组成(在txt里面写,然后另存为fasta文件就可以了)第一行是头部,第二行是序列,例如:
>1AKHA
KKEKSPKGKSSISPQARAFLEEVFRRKQSLNSKEKEEVAKKCGITPLQVRVWFINKRMRSK
-db后面是数据库文件位置。
-num_iterations后面是迭代次数,一般是3。
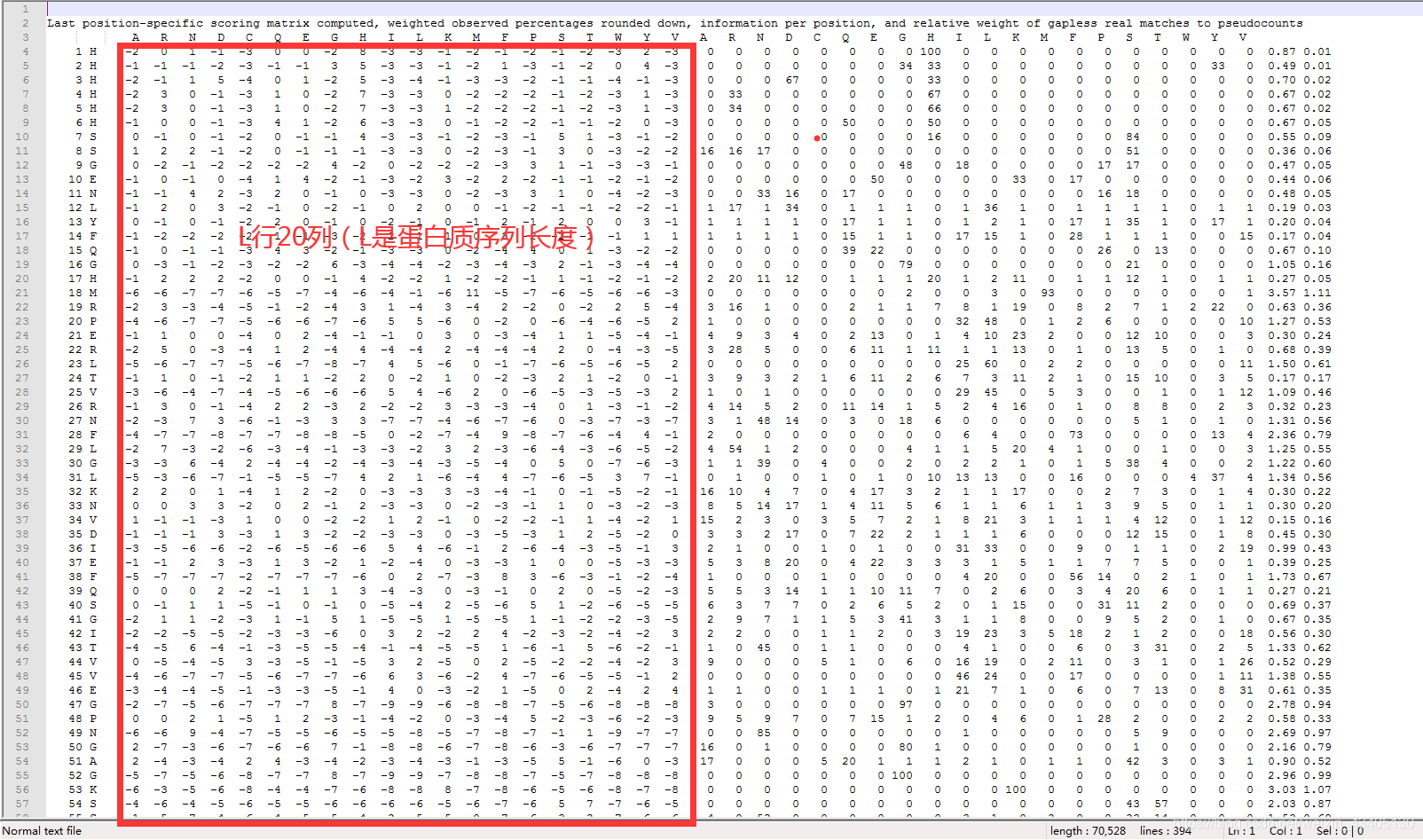
-out_ascii_pssm后面是输入的pssm文件路径,输出的pssm文件可以用Notepad++打开,如下图所示,红框里就是我们需要的数据。
没有报Error错误就说明成功了。
具体使用可以psiblast -help和BLAST ®命令行应用程序用户手册(美国生物技术信息中心)自行了解。
4.脚本
为了方便使用,我写了一个python脚本,可以参考一下
############################分割线##############################################
-- coding: utf-8 --
“”"
Created on Fri Jan 3 13:12:33 2020
获得pssm矩阵
@author: QYQ
“”"
import numpy as np
import os
class PSSM:
def __init__(self , db_filepath , input_filepath , output_filepath):#文件路径输入记得加r
assert os.path.isfile(db_filepath),'数据库文件位置不存在'
assert os.path.isfile(input_filepath),'输入文件位置不存在'
assert os.path.isfile(output_filepath),'输出文件位置不存在'
self.__db_filepath = db_filepath
self.__input_filepath = input_filepath
self.__output_filepath = output_filepath
def getSequence(self):#序列处理
return self.__Sequence
def setSequence(self,Sequence):
'''
把序列(字符串)写入输入文件
会自动清除fasta文件内容,然后写入,头可以忽略
Sequence序列不区分大小写,没有要求,不符合规范的程序会自己忽略掉
'''
self.__Sequence = Sequence
content = '>qyq' + '\n' + Sequence
with open(self.__input_filepath , 'w+') as f:
read_date = f.read()
f.seek(0)
f.truncate()
f.write(content)
f.close()
def get_line_numpy( self , line ,start=11 , end=91 , step=4):
'''
输入一行,返回处理后的np数组
默认start是11,end是91,step是4
start=10 end=69 step=3
'''
np_line = np.zeros([20])
index = 0
for symbol , num in zip(line[start:end:step] , line[start+1:end:step]):
if symbol == '-' :
statu = -1
else:
statu = 1
np_line[index] = statu*int(num)
index = index + 1
return np_line
def getPSSM(self):
with open(self.__output_filepath , 'r') as f:
for i in range(2):#前2行无用
f.readline()
line = f.readline()#第三行判断
if line[12] == 'A':
line = f.readline()
pssm_np = self.get_line_numpy( line )
line = f.readline()
while line != '\n' :
pssm_np = np.vstack( [pssm_np , self.get_line_numpy( line )] )
line = f.readline()
else:
line = f.readline()
pssm_np = self.get_line_numpy( line ,10,69,3)
line = f.readline()
while line != '\n' :
pssm_np = np.vstack( [pssm_np , self.get_line_numpy( line ,10,69,3)] )
line = f.readline()
f.close()
return pssm_np
def setPSSM(self):
'''
生成pssm文件
返回0 说明成功,返回1说明失败
软件生成pssm会自动重写文件内容
'''
#psiblast -in_msa D:\NCBI\input\test.fasta -db D:\NCBI\blast-2.3.0+\db\uniprot-all.fasta -comp_based_stats 1 -inclusion_ethresh 0.001 -num_iterations 3 -out_ascii_pssm D:\NCBI\output\1.pssm
#第二个序列ATCAATATCCACCTGCAGATTCTACCAAAAGTGTATTTGGAAACTGCTCCATCAAAAGGCATGTTCAGCTGAATTCAGCTGAACATGCCTTTTGATGGAGCAGTTTCCAAATACACTTTTGGTAGAATCTGCAGGTGGATATTGAT跑不出来
cmd = 'psiblast -in_msa '+self.__input_filepath + ' -db ' + self.__db_filepath +r' -comp_based_stats 1 -inclusion_ethresh 0.001 -num_iterations 3 -out_ascii_pssm ' + self.__output_filepath
statu = os.system(cmd)
assert statu == 0 , '生成PSSM文件失败,cmd命令有错'
if name == “main”:
pssm = PSSM(r’D:\NCBI\blast-2.3.0+\db\uniprot-all.fasta’,r’D:\NCBI\input\test.fasta’,r’D:\NCBI\output\1.pssm’)
Se = ‘ATCAATATCCACCTGCAGATTCTACCAAAAGTGTATTTGGAAACTGCTCCATCAAAAGGCATGTTCAGCTGAATTCAGCTGAACATGCCTTTTGATGGAGCAGTTTCCAAATACACTTTTGGTAGAATCTGCAGGTGGATATTGAT’
pssm.setSequence(Se)#写入序列
pssm.setPSSM()#生成pssm文件
pssm1 = pssm.getPSSM()#获得pssm矩阵
##################################分割线#####################################
注意:排版有误,分割县内都是代码,所以复制黏贴后还需自己改。有些序列会报告错误,看Error的错误信息可以自行解决。如果出现下图,我的解决办法是把结果相同(比如都是结晶蛋白,看你自己的分类问题而定)的序列替换掉。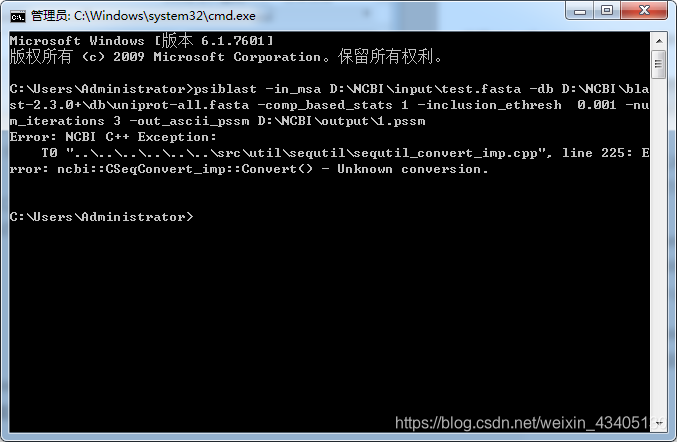
Last Update: 2021/9/7