mirnaDetect是使用java语言编写的用来在大规模基因数据中挖掘潜在的前体miRNAs的软件包。程序基于搜索和机器学习算法,mirnaDetect目前只支持Windows操作系统。
输入:fasta格式的基因序列文件
输出:查询序列中潜在的pre-miRNAs结果,gff3格式结果
下载解压mirnaDetect
首先使用软件mirnaDetect对序列注释,运行如下:
1,将测试文件test.txt文件拷贝到blast目录下,在doc中进入blast目录:
(1)formatdb.exe -i test.txt -p F -o T
(2)blastall.exe -p blastn -i mature.fa -d test.txt -o test_blastout.txt -e 0.005
注意:“test_blastout.txt”是BLAST结果文件;“-e 0.005”用来调整一个匹配的alignment质量。
2,把test.txt和test_blastout.txt文件拷贝到mirnaDetect目录下,在doc中进入mirnaDetect目录:
java -jar mirnaDetect.jar test.txt test_blastout.txt 0.5 4
注意“0.5”代表libSVM的分类阀值(从0到1),阀值越高,注释到的pre-miRNA越少,但是置信度越高;outlast.txt为结果文件。
3,如果需?蒰ff3文件,在上面两步后:
java -jar createGff3.jar outlast_info.txt outlast_simple.txt gff3_out.txt
注意:“gff3_out.txt”即为gff3格式结果文件。
示例(以下载的文件包中testData里的test.txt文件为例):
将test.txt文件拷贝到blast目录下
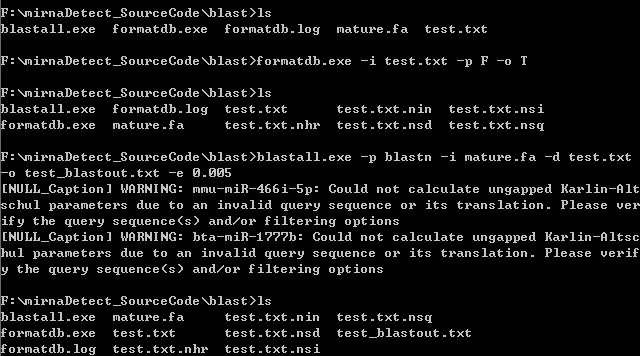
test_blastout.txt是BLAST结果文件
2,把test.txt和test_blastout.txt文件拷贝到mirnaDetect目录下,在doc中进入mirnaDetect目录
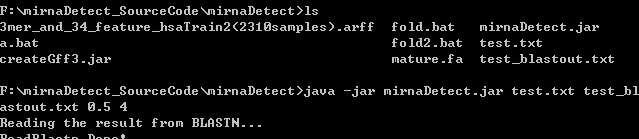

outlast.txt为结果文件
3,如果需要生成gff3文件,在上面两步后
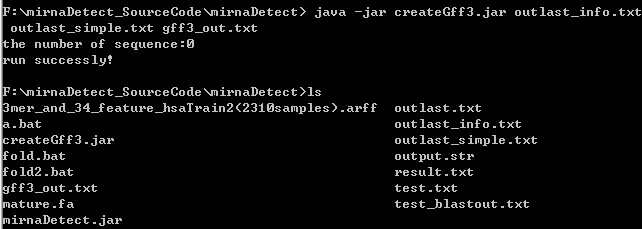
gff3_out.txt即为gff3格式结果文件
|